羊肚菌Morchella spp.是子囊菌门Ascomycota、盘菌纲Pezizomycetes、羊肚菌属Morchella真菌的统称,是国际上公认的名贵食用菌,分布于世界各地尤其是北温带地区(O'Donnell et al. 2011;Du et al. 2012;Kuo et al. 2012;刘伟等 2017a)。Ower(1982),Ower et al.(1986)首次成功实现其室内栽培,随后相继发布了详尽的羊肚菌室内栽培专利,由于羊肚菌生物学基础认知的匮乏,该栽培技术并得到很好的推广(Ower et al. 1986;刘伟等 2017a)。我国羊肚菌栽培技术在外源营养袋成功应用和易出菇品种的成功选育后得以快速发展,栽培区域遍布除热带地区外的全国各地;然而每年近70%的从业者无法稳定盈利,其主要原因是羊肚菌生物学基础薄弱,如育种理论薄弱、优良品种的评价与保持体系缺乏、菌种老化和退化明显等(刘伟等 2017b;赵永昌等 2018)。
双亲间DNA序列的多态性是发展分子标记的基础,基于PCR的共显性分子标记是最常用的标记类型,如单核苷酸多态性(single nucleotide polymorphism)SNP标记和插入缺失长度多态性(insertion-deletion length polymorphism)Indel标记(Powell et al. 1996;Bond et al. 1998;Vignal et al. 2002;Väli et al. 2008)。SNP和Indel是多态性比率较高的分子标记,水稻基因组中约几百bp的序列长度就有1个SNP或Indel(Alexandrov et al. 2014),而人类基因组中平均1.9kbp中有1个SNP(Group 2001),这些标记在图位克隆、分子标记辅助育种方面的研究受到越来越多的重视(Eathington et al. 2007)。
早期的羊肚菌属分子标记研究主要基于随机性标记如RAPD(Singh et al. 2004;Dalgleish & Jacobson 2005;刘伟等 2018b)、AFLP(Pagliaccia et al. 2011;陈立佼等 2013)、RFLP(Bunyard et al. 1994,1995)和ISSR(刘文丛等 2011)等对属内或种内群体进行遗传学研究;陈立佼等(2013)利用AFLP标记对尖顶羊肚菌M. conica的组织分离和单孢群体进行了分析指出,是否产菌核和产菌斑的形态特征和群体多态性具有一致性;刘伟等(2018)用RAPD分子标记对中国的36份羊肚菌栽培种植资源进行了评价,结果显示RAPD可以很容易将供试的栽培菌株划分为梯棱羊肚菌M. importuna和六妹羊肚菌M. sextelata两个类群,并针对特定菌株开发了RAPD-SCAR特征性标记。基因组测序技术加深了我们对物种的理解,也为我们开发相关的标记提供便利,我们早期基于粗柄羊肚菌M. crassipes的转录组数据对其SSR序列特征进行过初步分析(刘伟等 2017c)。目前还未有关于羊肚菌SNP、Indel标记的开发研究。
影像学、分子遗传学、基因组学和栽培学证据证实梯棱羊肚菌的异宗结合生活史秉性,两种不同极性的菌株配合是完成有性生活史所必须的条件(Chai et al. 2017;Du et al. 2017;He et al. 2017;Liu et al. 2018b;刘伟等 2019)。基因组测序技术的快速发展和费用的大幅度降低,基于基因组数据进行羊肚菌的遗传连锁图谱的构建、图位克隆和后期的分子标记辅助育种成为可能。本文基于课题组早期的基因组数据,对梯棱羊肚菌全基因组范围内SNP/Indel进行分析,并对变异位点序列长度≥11bp的Indel标记进行了开发,通过原生质体单细胞分离技术获得栽培品种M04的2个可亲和的同核体菌株,结合58个子囊孢子群体,初步构建了梯棱羊肚菌的遗传连锁图谱。
1 材料与方法
1.1 菌株群体构建
1.1.1 菌株:梯棱羊肚菌4号商业栽培菌株(M04)为野生菌株的自然选育驯化而得,保存于华中农业大学应用真菌研究所菌种保藏中心。
1.1.2 作图群体的构建:通过显微操作对一个成熟子囊果弹射的孢子进行挑取、萌发后获得,通过交配型基因特征性引物Mat1-1-1TF/R和Mat1-2-1TF/R对单孢菌株进行同核体的验证,单孢菌株记为M04Mn,n为第n个单孢(刘伟等 2019)。
1.1.3 M04同核体菌株制备:采用原生质体单细胞分离获得,简要操作如下:M04保藏菌株活化后,置于CYM液体培养基培养2-3d得到幼嫩菌丝,用0.6mol/L甘露醇作为渗透剂,2%的溶壁酶(广东微生物研究所)30℃酶解4h,无菌棉柱过滤去除菌丝碎片得到原生质体,经洗涤定量后,按照约102个原生质的浓度涂布至再生培养基(含有0.6mol/L甘露醇的CYM固体培养基),23℃倒置培养2-3d,随时进行观察,挑取萌发的单菌落作为待定菌株。用交配型基因特征性引物对待定的原生质体单细胞分离菌株进行交配型基因的判定。
1.2 DNA的制备
采用CYM完全培养基,9cm直径平皿铺无菌玻璃纸,接种后23℃避光培养,获得洁净的菌丝体。将菌丝体用液氮研磨后,用裂解液(CTAB 2%,PVP 2% Tris-HCl 100mmol/L,EDTA 20mmol/L,NaCl 1.4mol/L)裂解获得DNA粗提液,经体积比为25:24:1苯酚氯仿异戊醇去除蛋白后再经乙醇沉淀、RNase A消解,最终获得高质量DNA,稀释至50ng/μL,4℃储存备用。
1.3 SNP/Indel分析
梯棱羊肚菌M04两个单孢同核体菌株M04M24和M04M26的交配型基因分别是Mat1-2和Mat1-1,我们对这两个菌株进行了全基因组测序(Liu et al. 2018)。以梯棱羊肚菌M04M26菌株的基因组物理图谱为基准(NCBI基因组序列号:QOKS00000000),使用Trimmomatic-0.36将M04M24单孢菌株的基因组原始数据raw data进行低质量碱基的截断和过滤(M04M24:270bp PE150和800bp PE125 Illumina双末端测序,两个文库为4G原始测序数据;原始数据参考NCBI SRA数据库:SRR7448156-SRR7448157),得到clean data(Bolger et al. 2014);随后,使用Bowtie2设置不严格阈值(默认参数)将clean data比对到M04M26基因组上(Langmead & Salzberg 2012),利用Picard(http://picard.sourceforge.net/)软件去除PCR重复;其次,利用Bless(-kmer length 21)软件对去除了PCR重复的数据进行reads修正(Heo et al. 2014);再次使用Bowtie2,设置较严格阈值(--score-min L,-0.3,-0.3)将修正后的数据比对到基因组上;再利用Picard软件去除PCR重复,得到最终的可用于SNP/Indel分析的BAM文件。最后使用GATK的HaplotypeCaller命令对全基因组范围内的SNP/Indel进行分析,使用GATK的VariabtFiltration对结果进行过滤(QUAL<30.0;QD<13.0;MQ<20.0;FS>20.0;MQRankSum<-3.0;ReadPosRankSum<-3.0),最后借助SnpEff软件对获得SNP/Indel进行注释(McKenna et al. 2010;Cingolani et al. 2012)。
1.4 Indel标记的设计及扩增分析
选择序列长度≥11bp的Indel作为待开发标记位点,按照通用的引物设计原则,通过Primer3(http://primer3.sourceforge.net)对获得的Indel进行批量引物设计:上下游引物分别落在Indel位点的两端,引物的长度(20±2)bp,退火温度设定(60±3)℃,引物的GC% 30%-70%,目的片段长度100-250bp。Indel标记引物的命名按照“Scaffold编号”+“_”+“变异位点在基因组上的实际位置”,如20_504808引物代表第20个Scaffold上变异位点在504 808bp位置的Indel引物。
Indel扩增反应体系:5μL 2×Taq PCR MasterMix,2.5μmol/L上下游引物各0.5μL,50ng/μL DNA模板0.5μL,用ddH2O补足到10μL终体积。扩增条件预变性95℃ 3min,之后95℃ 15s,60℃ 15s,72℃ 25s 35个循环,循环结束后72℃ 5min补平。变异位点大于12bp的扩增产物通过3%琼脂糖凝胶电泳检测,变异位点小于12bp的扩增产物用6%尿素变性PAGE电泳,印染后拍照并统计(McKenna et al. 2010)。
1.5 梯棱羊肚菌遗传连锁图谱的构建
将统计的多态性条带数据类型定为DH类型,与M04P01基因型一致的记为“a”,与M04P40一致的记为“b”,杂合型条带记为“-”,遗传连锁图使用JoinMap®3.0软件构建,分子标记多态性按软件推荐方法读取,采用卡方(χ2)检验标记是否符合1:1分离,以LOD 2.0-10.0构建连锁群,以0.45作为最大重组值,用Kasambi方法换算成图距(Voorrips 2002;Alwala et al. 2008)。
2 结果与分析
2.1 梯棱羊肚菌基因组范围SNP/Indel的响应
梯棱羊肚菌M04的两个同核菌株M04M24和M04M26之间共计鉴定到17 104个SNP位点和1 334个Indel位点,平均每M碱基序列长度的位点变异数量为361个。2-10bp的插入缺失Indel标记1 147个,最大的插入缺失发生在Scaffold06染色体上547 436位置的117bp片段缺失。根据SNP/Indel的注释可知,大部分的SNP/Indel(73.4%)位于基因间隔区域,基因的上游和下游SNP/Indel发生数量基本相当,占比13.95%-14.02%,有9.14%位于内含子区域,这些变异位点对基因的功能影响可能不大;外显子区域总计检测到3 042个变异位点,占总数的16.50%,对基因功能产生确定影响的移码突变有1 088个,占5.90%,错义突变有916个,占比4.97%;另外有5.10%(940个)的同义突变(表1)。
表1 梯棱羊肚菌全基因组范围SNP/Indel特征信息
Table 1
| 条目 Type | 数量 Count | 占比 Percent (%) |
|---|---|---|
| 有效基因组长度 Genome effective length (bp) | 51 065 045 | |
| 变异位点总数 Number of variants (n) | 18 438 | |
| 单核苷酸多态性SNPs (n) | 17 104 | |
| SNP频率SNP frequency (SNPs/Mbp) | 361 | |
| 插入缺失多态性Indel (n) | 1 334 | |
| 基因上游区域Upstream (n) | 2 572 | 13.95 |
| 基因下游区域Downstream (n) | 2 585 | 14.02 |
| 基因间隔区Intergenic (n) | 13 533 | 73.40 |
| 外显子Exon (n) | 3 042 | 16.50 |
| 内含子Intron (n) | 1 686 | 9.14 |
| 移码突变Frameshift_variant (n) | 1 088 | 5.90 |
| 错义突变Missense_variant (n) | 916 | 4.97 |
| 同义突变Synonymous_variant (n) | 940 | 5.10 |
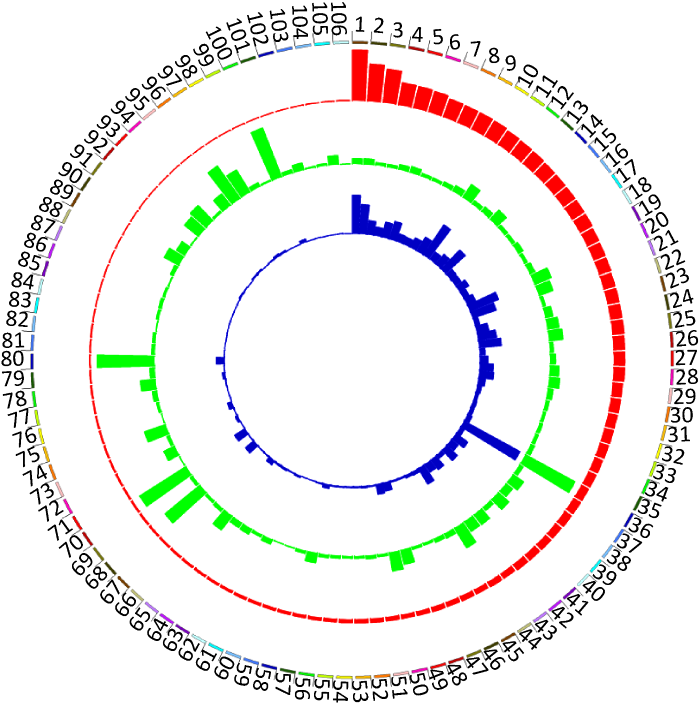
不同Scaffold上的SNP/Indel出现频率不同,SNP频率最大的为Scaffold80和Scaffold36,平均每M碱基分别包含2 856和2 811个SNP,59个Scaffold上的SNP频率超过200个/Mbp,占总SNP变异位点的86%(图1);SNP频率较低的有9个Scaffold,每M碱基少于100个,如Scaffold60和Scaffold75两条序列长度分别为184 065bp和98 078bp,只检测到3个SNPs,这些Scaffold是否具有保守的功能结构域有待进一步研究。
图1
图1
梯棱羊肚菌染色体水平的SNP分布及频率
最外圈:染色体编号;红色内圈:染色体的长度;绿色内圈:SNPs频率;蓝色内圈:不同染色体上的SNP数量
Fig. 1
The SNP distribution and frequency at chromosome level of Morchella importuna.
The outer circle: Chromosome number; The red circle: Length of the chromosomes; The inward green circle: The SNPs frequency; The inner blue circle: Number of SNPs on corresponding different chromosomes.
2.2 Indel标记的开发与群体扩增
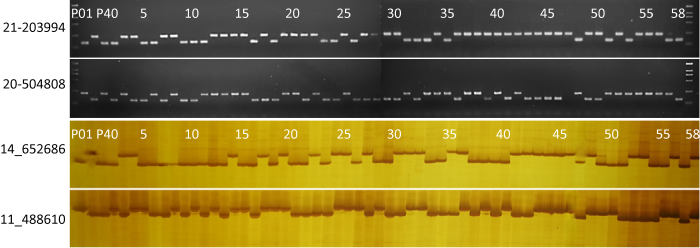
变异位点序列长度≥11bp的片段共计176个,其中121个片段为缺失,55个片段插入,基于这些变异位点设计Indel引物并进行扩增验证,149对可以成功扩增,其中75对引物具有多态性,多态性比率50.3%。在多态性检测方面,当变异位点序列长度大于等于13bp的时候,即可用3%琼脂糖凝胶电泳进行检测,而再小的片段则需要分辨率更高的变性PAGE进行检测(图2)。
图2
图2
梯棱羊肚菌Indel引物的多态性扩增
Fig. 2
Polymorphism amplification of Indel primers in the mapping population of Morchella importuna.
2.3 原生质体单细胞分离和单孢群体的制备
对梯棱羊肚菌M04菌株进行原生质体单细胞分离分析,共计挑取82个单菌落分离株,交配型结构分析显示,其中37个分离株同时含有Mat1-1-1和Mat1-2-1两种基因片段,它们可能是不同原生质体再生的混合体;45个单菌落为同核体,单细胞比例占总挑取菌落数量的54.3%。45个同核体菌株中,其中含有Mat1-1-1基因型的菌株38株,含有Mat1-2-1基因型的菌株7株,两者比例5.43:1,原生质体单细胞群体中,严重偏向于Mat1-1-1基因型。
在M04的自然弹射的子囊孢子印中,通过显微操作,共计挑取65个单子囊孢子,成功萌发58个单菌落,萌发率至少89%以上,这与羊肚菌子囊孢子萌发率的研究一致,羊肚菌子囊孢子即便在多年的保存之后仍可以具有较高的萌发率。58个单孢群体经交配型基因验证显示,所有的单孢中只含有两种交配型基因中的一种,其中Mat1-1-1基因型菌株25株,Mat1-2-1基因型33株,卡平方检验结果(χ2=1.10<χ20.05=3.84),两种交配型分离符合1:1。
2.4 遗传连锁图谱的构建
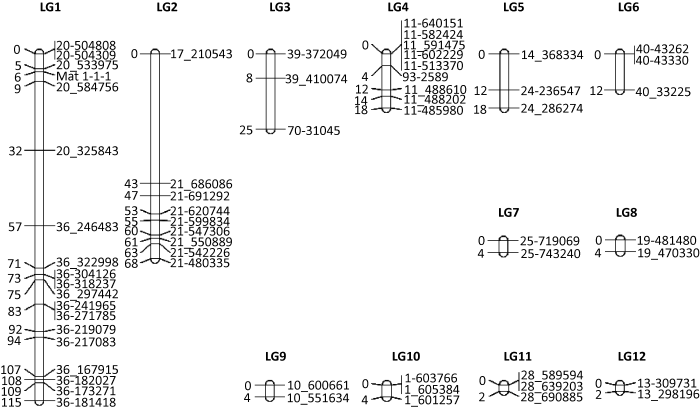
经筛选选定75个Indel标记在作图群体中具有多态性,连同交配型基因片段,共计76个标记用于连锁群的构建,剔除不连锁条带位点后,连锁结果显示60个条带位点分布在12个连锁群上。连锁群的总长度约为273.7cM(图3)。交配型基因位于LG1连锁群上,与临近的标记20_533975和20_584756的遗传距离分别为0.138cM和3.565cM,这与Mat1-1交配型基因的物理图谱在两者之间相一致(Mat1-1基因座:Scaffold020:537106-566670)。最大的连锁群LG1包含18个Indel标记和1个交配型位点基因,总长度114.709cM,标记之间平均距离6.04cM。
图3
从连锁图谱上可以看出,来自同一个Scaffold的遗传标记连锁在同一个连锁群上,物理序列位置和遗传图谱基本相互对应,符合预期结果,如LG1连锁群上,Scaffold36上的13个多态性Indel标记连锁在连锁群的一端,整体呈顺序排列,覆盖的基因组序列总长度约155kb。有10个不同的Scaffold分别连锁在5个最大的连锁群,如Scaffold20和Scaffold36连锁在LG1,Scaffold17和Scaffold21连锁在LG2上,代表着这些Scaffold在物理图谱中应该为相连的序列,说明遗传连锁图谱可以对基因组组装起到辅助作用。由于本文使用到的Indel标记数量并未能全覆盖所有的Scaffold以及标记数量的限制,连锁群的总长度和标记密度偏低,实际的遗传连锁群数量有待进一步证实。
3 讨论
3.1 基因组范围内SNP/Indel变异位点的分析
基因组测序数据可以方便地进行全基因组范围内SNP/Indel分析和开发,在SNP/Indel挖掘方面,原始测序数据比对到基因组上的BAM文件准确性对SNP/Indel的响应有较大的影响,因此需要对常规的基因组重测序数据进行质控和比对参数的优化,尽可能的降低假阳性结果。本文总计在梯棱羊肚菌基因组内检测到18 438个SNP/Indel变异位点,平均每M碱基361个,略低于丝状真菌中的SNP频率。Payen et al.(2015)对7个块菌品种全基因组SNP分析显示,块菌的SNP频率为3 540SNPs/Mbp;10个Coccidioides posadasii和10个C. immitis的全基因组中的SNP频率分别为9 277SNPs/Mbp和5 251SNPs/Mbp(Neafsey et al. 2010);48个粗糙脉孢霉Neurospora crassa的基因组分析显示其SNP频率为3 375SNPs/Mbp(Ellison et al. 2011);均高于本研究的SNP频率,这可能与用于分析的材料选择和数量以及数据质控有关,本研究中选用的是两个来自于同一个子囊果的经过减数分裂的两个同核体单孢菌株,其遗传背景较近,同时设置了严格的SNP分析流程。
3.2 梯棱羊肚菌原生质体单细胞分离及同核体交配型偏分离
梯棱羊肚菌异宗结合生活史类型已得以证实,本文通过原生质体单细胞分离技术,从M04菌株获得两个不同交配型的可亲和同核体菌株,两种交配型比例明显偏离1:1(Mat1-1-1基因型:Mat1-2-1基因型为38:7)。这种现象与其他食用菌类似,例如,两个斑玉蕈Hypsizygus marmoreus品种的原生质体单核化比例为29.7%和33.1%,两种A1B1和A2B2的交配型比例也呈明显的偏分离现象,分别达91:11和113:21,不符合1:1的分离比(潘越等 2014);大球盖菇Stropharia rugosoannulata的原生质体再生群体中单核和双核的比例为2.9:1,单核化比例达77.6%,其交配型比例存在偏离的现象(Yan et al. 2004)。香菇Lentinula edodes中也存在这种明显的偏分离现象(Fukumasa-nakai et al. 1994;朱朝辉等 2000),偏分析现象可能与不同交配型基因所代表的核型在再生能力方面存在着差异有关。
3.3 Indel分子标记与遗传连锁图谱
Indel分子标记具有稳定性好、共显性、通用性的特点,被认为是比较可靠的分子标记, Indel标记的开发主要基于测序数据获得,近年来基因组测序、重测序工作的开展为该标记的开发提供了便利(Ajawatanawong & Baldauf 2013)。遗传连锁图谱是依据基因的连锁、重组交换值构建的图谱,展现的是基因或遗传标记在染色体上的相对位置,是基因组研究中的一个重要组成部分,也是分子遗传育种的基础,可为进一步的基因功能分析、基因组组装提供帮助(刘伟等 2010;Foulongne-Oriol 2012)。随着测序技术的普及,基于基因组序列开发的SSR、STS、SNP、Indel标记应用越来越多(Labbé et al. 2008;Foulongne-Oriol 2012;Gong et al. 2014)。本文基于全基因组水平的SNP/Indel变异位点的分析,对≥11bp长度的变异位点进行Indel标记的开发,在梯棱羊肚菌群体内进行了扩增分析和遗传连锁图的构建。由于分子标记数量有限,所构建的遗传连锁图谱无论是图谱总长度还是平均密度上均显不足,后期将在此基础上进一步通过标记数量的增加完善梯棱羊肚菌遗传连锁图谱的构建,为将来的基因组染色体水平的拼接、图位克隆、分子标记辅助育种打下基础。
参考文献
Evolution of protein indels in plants, animals and fungi
SNP-seek database of SNPs derived from 3000 rice genomes
Linkage mapping and genome analysis in a Saccharum interspecific cross using AFLP, SRAP and TRAP markers
Trimmomatic: a flexible trimmer for Illumina sequence data
Single-nucleotide polymorphism in the human mu opioid receptor gene alters β-endorphin binding and activity: possible implications for opiate addiction
A systematic assessment of Morchella using RFLP analysis of the 28S ribosomal RNA gene
Phylogenetic resolution of Morchella, Verpa, and Disciotis [Pezizales: Morchellaceae] based on restriction enzyme analysis of the 28S ribosomal RNA gene
Characterization of mating-type idiomorphs suggests that Morchella importuna, Mel-20 and M. sextelata are heterothallic
Genetic diversity among Morchella conica based on AFLP analysis
A program for annotating and predicting the effects of single nucleotide polymorphisms, SnpEff: SNPs in the genome of Drosophila melanogaster strain w1118; iso-2; iso-3
A first assessment of genetic variation among Morchella esculenta (Morel) populations
Multigene molecular phylogenetics reveals true morels (Morchella) are especially species-rich in China
Mixed-reproductive strategies, competitive mating-type distribution and life cycle of fourteen black morel species
Molecular markers in a commercial breeding program
Population genomics and local adaptation in wild isolates of a model microbial eukaryote
Genetic linkage mapping in fungi: current state, applications, and future trends
Dedikaryotization of the shiitake mushroom, Lentinula edodes by the protoplast regeneration method
Constructing a new integrated genetic linkage map and mapping quantitative trait loci for vegetative mycelium growth rate in Lentinula edodes
A map of human genome sequence variation containing 1.42 million single nucleotide polymorphisms
Live cell confocal laser imaging studies on the nuclear behavior during meiosis and ascosporogenesis in Morchella importuna under artificial cultivation
BLESS: bloom filter-based error correction solution for high-throughput sequencing reads
Taxonomic revision of true morels (Morchella) in Canada and the United States
A genetic linkage map for the ectomycorrhizal fungus Laccaria bicolor and its alignment to the whole‐genome sequence assemblies
Fast gapped-read alignment with Bowtie 2
c. Distribution and sequence characteristics of SSR in transcriptome of Morchella crassipes
Cultivation of monosporic and hybrid populations and polarity analysis of Morchella importuna
Opposite polarity monospore genome de novo sequencing and comparative analysis reveal the possible heterothallic life cycle of Morchella importuna
Genetic diversity analyses and species specificity RAPD-SCAR marker exploitation of the cultivated Morchella strains
Research advance of molecular genetic linkage map in macrofungi
b. The current situation and trend of the development of Morchella industry in China
Genetic diversity of five species morchella from northwest Yunnan Province revealed by ISSR analysis
The Genome Analysis Toolkit: A MapReduce framework for analyzing next-generation DNA sequencing data
Population genomic sequencing of Coccidioides fungi reveals recent hybridization and transposon control
Phylogeny and historical biogeography of true morels (Morchella) reveals an early Cretaceous origin and high continental endemism and provincialism in the Holarctic
Notes on the development of the morel ascocarp: Morchella esculenta
Cultivation of Morchella
Development of molecular markers and preliminary investigation of the population structure and mating system in one lineage of black morel (Morchella elata) in the Pacific Northwestern USA
Variance analysis of different mating type strains in mononuclear protoplast of Hypsizzygus marmoreus
A survey of genome‐wide single nucleotide polymorphisms through genome resequencing in the Périgord black truffle (Tuber melanosporum Vittad.)
Polymorphism revealed by simple sequence repeats
Arbitrary primer based RAPD - a useful genetic marker for species identification in morels
Insertion-deletion polymorphisms (indels) as genetic markers in natural populations
A review on SNP and other types of molecular markers and their use in animal genetics
MapChart: software for the graphical presentation of linkage maps and QTLs
Characterization of protoplasts prepared from the edible fungus, Stropharia rugoso-annulata
Rational understanding of many problems in the development of Morchella industry
Study on the relationship between protoplasted monokaryon regeneration and mating types of Lentinula edodes
尖顶羊肚菌遗传多样性的AFLP分析
c. 粗柄羊肚菌转录组的SSR分布和序列特征分析
梯棱羊肚菌单孢及杂交群体的栽培出菇试验和极性分析
羊肚菌栽培菌株遗传多样性分析及种特异性RAPD-SCAR标记开发
大型真菌分子遗传连锁图谱研究进展
b. 我国羊肚菌产业发展的现状及趋势
滇西北地区5种羊肚菌遗传多样性的ISSR分析
斑玉蕈不同交配型的原生质体单核菌株差异分析
理性认识羊肚菌产业发展诸多问题
香菇原生质体单核体的再生与交配型的关系